Bändermodell (Proteine)
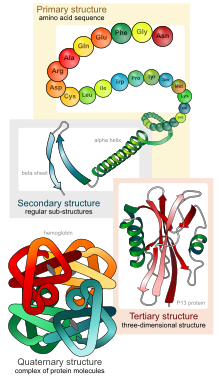
Proteinstrukturen, von der primären zur quartären Struktur.
Bändermodelle sind Molekülmodelle von Proteinstrukturen. Sie visualisieren Elemente der Sekundärstruktur wie eine α-Helix oder ein β-Faltblatt und erlauben eine Darstellung der Tertiärstruktur. Verschiedene Polypeptide können zur besseren Unterscheidung farblich unterschiedlich markiert sein. Bändermodelle werden bei der molekularen Modellierung eingesetzt, z. B. beim Protein-Engineering. Das Bändermodell wurde 1980/81 durch Jane Richardson etabliert. Ähnliche Ansätze gab es aber schon früher.
Strukturdarstellungen
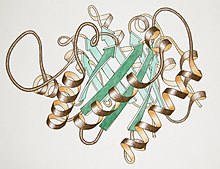
Triosephosphatisomerase, handgezeichnet von Jane Richardson.
| Sekundärstruktur | |
|---|---|
| α-Helices | Zylindrische spiralförmige Bänder, mit der Bänderebene ungefähr auf der Peptidbindungsebene. |
| β-Stränge | Pfeile mit einer Dicke von 25 % der Breite in Richtung von N- zu C-Terminus. β-Faltblätter bestehen aus parallel angeordneten β-Strängen mit β-Schleifen. |
| Schleifen und andere | |
| Nichtrepetitive Schleifen | Runde Stränge entlang der α-C-Atome der Aminosäuren, die vom Vordergrund zum Hintergrund dünner werden. |
| Verbindungen zwischen Schleifen und Helices | Runde Stränge entlang der α-C-Atome der Aminosäuren, die beim Übergang zur α-Helix flacher werden. |
| Andere Merkmale | |
| N- und C-Terminus | Kleine Pfeile an einem oder beiden Enden oder mit Buchstaben codiert. Für β-Stränge ist die Richtung des Pfeils ausreichend. Oftmals wird hierfür ein Farbgradient entlang der Aminosäuresequenz verwendet. |
| Disulfidbrücken | Mit verschränktem Doppel-S oder mit einem Zick-Zack-Strich |
| Prosthetische Gruppen oder Inhibitoren | Stabmodell, Kugel-und-Stabmodell |
| Metalle | Kugeln |
| Schattierung und Farbe | Der Kontrast nimmt zum Hintergrund hin ab |
Software
Verschiedene Programme zur Darstellung von Proteinen im Bändermodell wurden entwickelt.


© biancahoegel.de
Datum der letzten Änderung: Jena, den: 26.05. 2024